
Regulación de la expresión génica en Procariontes
- Regulación de la expresión génica en bacterias: el operón
• Sistemas constitutivos y sistemas adaptativos
• Sistemas inducibles y sistemas represibles
Videos interesantes |
• Control positivo y control negativo
• El operón lactosa: control negativo
• El operón lactosa: control positivo
• El operón triptófano: regulación por el atenuador.
Interacción entre operones
- Regulación de la expresión génica en fagos
• Regulación del fago LAMBDA.
-Regulación simultánea de genes situados a distancia. Respuesta SOS
-Síntesis coordinada de proteínas ribosómicas y ARNr.
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN BACTERIAS
En las bacterias, a pesar de ser organismos unicelulares, también es necesario regular la expresión de los genes adaptándola a las necesidades ambientales. Es un principio de economía celular el que la expresión de los genes esté regulada según las circunstancias celulares. Un buen ejemplo de esta situación en bacterias es la regulación de las enzimas implicadas en el metabolismo de los azúcares. Las bacterias pueden emplear para obtener energía distintas fuentes de carbono, como la glucosa, lactosa, galactosa, maltosa, ramnosa y xilosa. Existen enzimas capaces de introducir cada uno de estos azúcares en la bacteria y enzimas capaces de romperlos para obtener energía. Lógicamente, sería un despilfarro energético producir simultáneamente todos los enzimas necesarios para metabolizar los diferentes azúcares mencionados. Por consiguiente, sería mucho más económico para la célula producir solamente las enzimas necesarias en cada momento, es decir, si en el medio en el que vive la bacteria la principal fuente de carbono es la lactosa, solamente se expresarían los genes necesarios para metabolizar la lactosa, mientras que los otros genes no se expresarían. Por tanto, es esencial que exista un mecanismo de regulación de la expresión génica, de manera que los genes se expresen cuando sea necesario.
La regulación de la producción de proteínas (síntesis de proteínas) considerando el proceso en su conjunto, puede llevarse a cabo en tres niveles:
De los tres niveles de regulación, uno de los mejor conocidos actualmente es la regulación durante la transcripción. Aunque la regulación de la transcripción en eucariontes es más compleja que en bacterias, muchos de sus aspectos son similares. Por tanto, comenzaremos por el estudio de la regulación de la transcripción en bacterias.
SISTEMAS CONSTITUTIVOS Y SISTEMAS ADAPTATIVOS
Es evidente que existen algunos procesos metabólicos que son necesarios para el funcionamiento normal de casi todas las células, de manera que existen una serie de necesidades básicas para el mantenimiento normal de una célula. Por consiguiente, los genes que codifican para las enzimas necesarias para el metabolismo básico celular se están expresando continuamente, es decir, se expresan de forma constitutiva o continua. Por tal motivo, a este tipo de genes se les denomina, " genes que guardan la casa " o genes constitutivos . Estos genes que se están expresando continuamente no significa que su actividad no esté regulada, simplemente están sometidos a un tipo de regulación diferente que hace que se estén expresando siempre. Los genes constitutivos codifican para sistemas enzimáticos constitutivos, que se necesitan siempre para la actividad normal de la célula.
Frente a los genes constitutivos, nos encontramos con los genes que se expresan solamente en determinadas situaciones y que, por consiguiente, codifican para enzimas que solamente se necesitan en momentos concretos. A este tipo de genes se les llama genes adaptativos y a las enzimas codificadas por ellos, sistemas enzimáticos adaptativos . Se denominan así pensando en que se expresan cuando la célula se adapta a una determinada situación ambiental. En algunos libros de texto se denomina a este tipo de genes, genes regulados , sin embargo, esta nomenclatura no es demasiado correcta, ya que parece que los únicos genes cuya expresión está regulada serían estos.
SISTEMAS INDUCIBLES Y SISTEMAS REPRESIBLES
Sistemas inducibles: cuando el sustrato sobre el que va actuar la enzima provoca la síntesis del enzima. Al efecto del sustrato se le denomina inducción positiva . Por ejemplo, en E. coli en ausencia de galactósido (sustrato) hay unas diez unidades de galactosidasa (enzima) por miligramo de materia seca, mientras que en presencia de galactósido se detectan hasta 10.000 unidades de galactosidasa por miligramo de materia seca. Al compuesto que desencadena la síntesis del enzima se le denomina Inductor .
Sistemas represibles: cuando el producto final de la reacción que cataliza el enzima impide la síntesis de la misma. Este fenómeno recibe el nombre de inducción negativa . Al compuesto que impide la síntesis del enzima se le denomina correpresor .
Los sistemas inducibles se corresponden a procesos catabólicos de degradación, por ejemplo, el operón lactosa, el operón arabinosa, el operón maltosa. Se trata de sistemas enzimáticos encargados de degradar la lactosa, arabinosa, maltosa, etc.
Los sistemas represibles se corresponden con procesos de síntesis o Anabolismo, por ejemplo el operón triptófano y el operón histina. Se trata de las rutas metabólicas que conducen a la síntesis de triptófano y síntesis de histidina.
CONTROL POSITIVO Y CONTROL NEGATIVO
Control positivo: Se dice que un sistema está bajo control positivo cuando el producto del gen regulador activa la expresión de los genes, actúa como un activador .
Control negativo: se dice que un sistema está bajo control negativo cuando el producto del gen regulador reprime o impide la expresión de los genes, actúa como un represor .
Proteínas que regulan el inicio de la transcripción:
• Factores de especificidad (s70, s32, s54).
La ARN polimerasa de procariotas es una proteína con diferentes subunidades: está compuesta por dos subunidades aßß'w y a ella se le une otra subunidad s. Sin la subunidad sigma no puede iniciar la transcripción. La subunidad s supone el factor de especificidad de la ARN polimerasa, ya que reconoce al promotor, y existen diferentes tipos de subunidades sigmas que serán capaces de reconocer a diferentes tipos de promotores. La más conocida es la subunidad s70, pero a veces aparecen otras subunidades como la s32, de manera que la ARN polimerasa reconoce promotores para originar proteínas que actúan para choque térmico, un ejemplo de estas proteínas serían las chaperonas. Otra subunidad que puede aparecer es la s54 cuya secuencia es significativamente distinta a la s70 y la transcripción de genes por la ARN polimerasa que contiene este tipo de subunidad sigma está regulada exclusivamente por activadores cuyos sitios de unión al DNA, denominados amplificadores, generalmente están localizados 80-160 pares de bases aguas arriba desde el sitio de inicio. Incluso cuando los amplificadores se encuentran a más de una kilobase de distancia respecto de un sitio de inicio, los activadores s54 pueden activar la transcripción.
• Represores:
Son proteínas que se unen a determinadas secuencias de ADN llamadas operadores. Estos represores en el caso de procariotas impide el avance de la ARN polimerasa de manera que inhibe o reprime la transcripción cuando se une al operador.
• Activadores:
Son proteínas que se une a una determinada secuencia de ADN y facilitan la unión de la polimerasa al promotor, son por tanto estimuladoras de la transcripción.
Otros factores que también intervienen en la regulación de la transcripción:
Señales moleculares: Son determinadas moléculas que se pueden unir al represor y lo que hacen es generar un cambio conformacional en ellas que facilita o inhibe la unión de ambos tipos de proteínas al ADN. Modulan la actividad tanto de los represores como de los activadores.

Francois Jacob |
Jacques Monod |
Jacob, Monod y colaboradores analizaron el sistema de la lactosa en E. coli , de manera que los resultados de sus estudios permitieron establecer el modelo genético del Operón que permite comprender como tiene lugar la regulación de la expresión génica en bacterias. Jacob y Monod recibieron en 1965 el Premio Nobel por estas investigaciones.
Un Operón es grupo de genes estructurales cuya expresión está regulada por los mismos elementos de control (promotor y operador) y genes reguladores.
Los principales elementos que constituyen un operón son los siguientes:
EL OPERÓN DE LA LACTOSA:
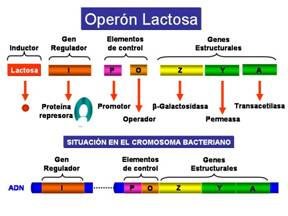
Estructura del operón de la lactosa:
1º Gen que codifica el represor, gen i, que tiene su propio promotor.
2º Sitio de unión para CRP
3º El promotor para los genes estructurales.
4º El operador
5º Genes estructurales. Son 3 y codifican para tres proteínas:
-Gen Z: codifica la ß-galactosidasa, esta enzima hidroliza la lactosa en glucosa y galactosa.
-Gen Y: codifica la lactosa permeasa que permite el transporte de lactosa al interior de la célula.
-Gen A: codifica la tiogalactosidotransacetilasa que facilita la detoxificación de algunos galactósidos que son tóxicos. No participa directamente en el metabolismo de la lactosa.
Gen regulador i:
codifica una proteína represora, la cual se une al operador impidiendo el avance de la ARN polimerasa y por tanto inhibe la transcripción. A esta proteína represora codificada por el gen i se le puede unir un inductor (que sería una señal molecular) que generaría un cambio conformacional en el represor impidiendo que se una al operador, por lo que en este caso sí tendría lugar la transcripción.
Inductores:
-Lactosa: No es un inductor muy potente.
- Alolactosa: Es un producto intermediario. Es el inductor más potente, pero el problema es que se degrada por la ß-galactosidasa.
-IPTG: (Isopropaniltiogalactosido). Es un compuesto químico artificial y es el más usado.
Represor:
Es una proteína tetramérica, formada por 4 subunidades idénticas entre sí. Cada subunidad diferente se llama protómero. Por tanto hay un solo protómero ya que las 4 son iguales. Hay simetría, la proteína es realmente un dímero de dímeros y cada dímero se une de forma independiente al ADN. Cada monómero está compuesto por 360 aminoácidos.
Cuando se une al operón no permite la expresión de los genes estructurales, debido a que impide el desplazamiento de la ADN polimerasa.
Estructura de cada monómero:
En cada uno se puede diferenciar a su vez un dominio de unión al ADN que es un dominio hélice-giro- hélice (en procariotas). Generalmente esta estructura está compuesta por unos 20 aminoácidos, en la que se pueden diferenciar 2 hélices a que forman un ángulo de 120º. La segunda de estas hélices es la que se une al surco mayor del ADN (es la que está en el extremo C terminal)
Operador: sitio de unión del represor
Realmente hay 3 regiones que desempeñan esta función:
O 1 ) Región de constituida por 35 pares de bases. De estos 35, 26 pb son simétricos y en esta región está incluido el punto de inicio de la transcripción. Se superpone con el sitio de inicio de la transcripción del gen Lac Z.
O 2 ) Está dentro del gen LacZ, situada aproximadamente unos 400 pb después del inicio de la transcripción.
O 3 ) Situada unos 82 pares antes del comienzo de la transcripción, al final del gen Lacl y el que codifica el represor.
En condiciones normales se
El represor se une de forma simultánea a dos regiones operadoras. Una siempre es la región O 1 , pero cuando esto pasa se forma una estructura en forma de bucle. Cuando se forma este bucle la ARNpol aunque parece que sí puede unirse al promotor no puede avanzar y la transcripción se reprime.
Si hay inductor en el medio éste se une a su región de unión (entre los dos dominios de unión al inductor, en la región central). Entonces se produce un cambio conformacional y las hélices de reconocimiento de cada uno de los monómeros varían su distancia entre ellas y no se pueden unir a dos surcos mayores consecutivos del ADN, de manera que no pueden ejercer su efecto represor.
EL OPERÓN LACTOSA: CONTROL NEGATIVO
El Operón lactosa, que abreviadamente se denomina Operón lac , es un sistema inducible que está bajo control negativo, de manera que la proteína reguladora, producto del gen regulador i , es un represor que impide la expresión de los genes estructurales en ausencia del inductor. El inductor del sistema es la lactosa. Como veremos más adelante, el operón lac también está bajo control positivo, ya que existe otra proteína que estimula la transcripción de los genes estructurales.
Los genes estructurales del operón lactosa son los siguientes:
El verdadero inductor del sistema es la Alolactosa y no la lactosa de manera que la ß-galactosidasa transforma la lactosa en Alolactosa . En los estudios del operón lactosa se utiliza como inductor un análogo sintético de la lactosa que es el Isopropil tiogalactósido (IPTG) . El IPTG no necesita ser transportado por la galactósido permeasa para entrar en la bacteria.
Las cepas normales de E. coli son inducibles, de manera que en ausencia del inductor (la lactosa), la proteína represora producto del gen i se encuentra unida a la región operadora e impide la unión de la ARN-polimerasa a la región promotora y, como consecuencia, no se transcriben los genes estructurales.
|
Operón lactosa en ausencia de lactosa |
Sin embargo, en presencia del inductor (la lactosa), este se une a la proteína reguladora que cambia su conformación y se suelta de la región operadora dejando acceso libre a la ARN-polimerasa para que se una a la región promotora y se transcriban los genes estructurales. Por consiguiente, la presencia del inductor hace que se expresen los genes estructurales del operón, necesarios para metabolizar la lactosa.
|
Operón lactosa en presencia de lactosa |
También es conveniente recordar que los tres genes estructurales del operón lactosa se transcriben juntos en un mismo ARNm, es decir que los ARN mensajeros de bacterias suelen ser policistrónicos, poligénicos o multigénicos . Sin embargo, en eucariontes los mensajeros suelen ser monocistrónicos o monogénicos , es decir, corresponden a la transcripción de un solo gen estructural.
OPERÓN LACTOSA: CONTROL POSITIVO
Como ya he mencionado anteriormente, el operón lactosa también está sujeto a un control de tipo positivo, de manera que existe una proteína que estimula la transcripción de los genes estructurales. En los sistemas de control negativo existe una proteína que que impide la transcripción de los genes estructurales, en los sistemas de control positivo existe una prteína activadora que estimula la transcripción de los genes. En principio existen cuatro tipos de sistemas posibles de regulación de la expresión génica:
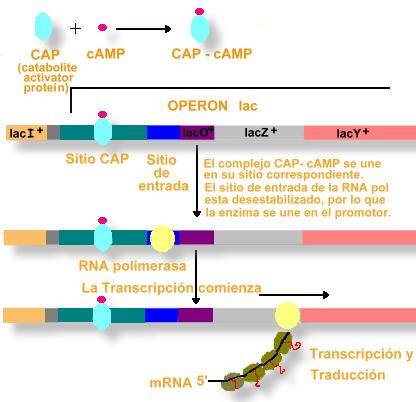
Por supuesto, un operón pude estar sujeto a más de un tipo de control, como sucede en el caso del operón lactosa que está bajo control negativo ejercido por la proteína represora y bajo control positivo ejecutado por una proteína activadora por catabolitos (CAP) también llamada proteína activadora del AMP cíclico (CRP) . El control positivo del operón lactosa como veremos está estrechamente relacionado con la Represión catabólica .
Cuando la bacteria E. coli crece en un medio que contiene glucosa, prefiere este azúcar como fuente de energía y como consecuencia los operones que ponen producen las enzimas necesarias para obtener energía de otros azúcares están bloqueados. Uno de los catabolitos del metabolismo de la glucosa actúa sobre el AMP cíclico (AMPc) . El AMP cíclico (AMPc) es necesario para la transcripción de todos los operones que son inhibidos por el catabolismo de la glucosa. Es decir, para que se transcriban los genes del operón lactosa se necesitan niveles elevados de AMPc. Esto mismo sucede con los operones de arabinosa, maltosa, galactosa, etc.. Se trata. por tanto de un sistema general de control positivo que se denomina represión catabólica . Cuando E. coli crece en un medio con glucosa, los niveles de AMPc son muy bajos y como consecuencia no se transcriben los operones de otros azúcares. Aún no se conoce el motivo por el que los niveles de AMPc son bajos cuando E. coli crece en un medio con glucosa como fuente de energía.
Cuando E. coli crece en un medio sin glucosa pero con lactosa los niveles de AMPc son altos, el AMPc se une a la proteína receptora de AMPc (CRP) activándola y la proteína activada CRP-AMPc a su vez estimula la transcripción de los genes estructurales del operón lactosa y de otros operones de azúcares. La proteína activadora por catabolitos (CAP) es un dímero que al unirse al AMPc se activa y estimula la transcripción de los genes del operón lactosa, de manera que la proteína activadora CAP-AMPc es necesaria para la unión de la ARN-polimerasa al promotor de los genes del operón lactosa. La proteína activadora CAP-AMPc parece ser que se une a la región promotora cerca del nucleótido que ocupa la posición -60 (60 nucleótidos antes del comienzo del gen z).
Los mutantes que afectan a la adenilato ciclasa, enzima que transforma el ATP en AMPc, muestran niveles muy bajos de expresión de los genes del operón lactosa, debido a que los niveles de AMPc son muy bajos en ausencia de glucosa.
Los mutantes que afectan a la proteína CRP o CAP también presentan niveles muy bajos de expresión de los genes del operón lactosa en ausencia de glucosa.
El operón triptófano (operón trp) es un sistema de tipo represible, ya que el aminoácido triptófano (Correpresor) impide la expresión de los genes necesarios para su propia síntesis cuando hay niveles elevados de triptófano. Sin embargo, en ausencia de triptófano o a niveles muy bajos se transcriben los genes del operón trp. Los elementos del operón trp son en esencia semejantes a los del operón lactosa:
En el siguiente esquema se indican los elementos del Operón Triptófano:
|
Elementos del Operón Triptófano |
Los genes estructurales del operón triptófano se encuentran en el mismo orden que actúan las productos codificados por ellos en la ruta biosintética del triptófano (ver el siguiente esquema).
|
Orden de los genes estructurales del operón triptófano y ruta de síntesis del triptófano |
En ausencia de triptófano, o cuando hay muy poco, la proteína reguladora producto del gen trpR no es capaz de unirse al operador de forma que la ARN-polimerasa puede unirse a la región promtora y se transcriben los genes del operón triptófano.
|
Operón triptófano: en ausencia de triptófano |
En presencia de triptófano, el triptófano se une a la proteína reguladora o represora cambiando su conformación, de manera que ahora si puede unirse a la región operadora y como consecuencia la ARN-polimerasa no puede unirse a la región promotora y no se transcriben los genes estructurales del operón trp.
|
Operón triptófano: en presencia de triptófano |
Por tanto, la diferencia esencial entre el operón lac (inducible) y el operón trp (represible), es que en este último el represor del triptófano solamente es capaz de unirse al operador cuando previamente está unido al trp. El operón del triptófano está siempre “conectado”, a no ser que se encuentre reprimido por la presencia de la molécula correpresora. Por el contrario el operón de la lactosa está “desconectado”, a no ser que sea inducido por una molécula inductora (alolactosa)
El represor del triptófano es un homodímero de dos subunidades idénticas de 108 aminoácidos cada una. Ambas subunidades que forman dicho dímero se une al ADN con motivos hélice-giro- hélice. El represor no se une por si mismo al operador trp. Para ello debe unirse a dos moléculas de triptófano, por lo que el represor solo actuará in vivo en presencia de triptófano.
Al estudiar más profundamente el operón trp se encontró que además del mecanismo de regulación represor-operador existía otro mecanismo de regulación que se denominó regulación por atenuación .
EL OPERÓN TRIPTÓFANO: REGULACIÓN POR ATENUACIÓN
Otro importante elemento de control del operón trp es la región atenuadora. Esta región proporciona a la ARN polimerasa una segunda oportunidad de regular la transcripción del operón del triptófano, en función de las necesidades celulares de los enzimas de la biosíntesis del triptófano.
|
La secuencia atenuadora se encuentra en la región líder
|
|
Secuencia de bases de la región atenuadora y comienzo de la secuencia del gen trpE |
El mecanismo de atenuación del operón trp utiliza señales en cuatro secuencias dentro de una región guía de 162 nucleótidos en el extremo 5' del ARNm que precede al codón de inicio del primer gen. Dentro de la secuencia guía se encuentra una región denominada atenuador, constituidas por las secuencias 3 y 4. Estas secuencias se aparean para formar una estructura en horquilla rica en G-C, seguida inmediatamente por una serie de residuos de uracilo. La estructura del atenuador actúa como terminador de la transcripción. La secuencia 2 es una secuencia alternativa para la secuencia 3. Si se aparean las secuencias 2 y 3, no se puede formar la estructura atenuadora y la transcripción continúa en los genes biosintéticos trp; el lazo formado por el apareamiento de las secuencias 2 y 3 no obstruye la transcripción.
La secuencia reguladora 1 es crucial para el mecanismo sensible al triptófano que determina si la secuencia 3 se complementa con la secuencia 2 (permitiendo que continúe la transcripción) o con la secuencia 4 (atenuando la transcripción). La formación de estructura atenuadora en horquilla depende de cómo transcurra la traducción de la secuencia 1 reguladora, que codifica un péptido guía (denominado así porque está codificado por la región guía del ARNm) de 14 aminoácidos, dos de los cuales son residuos de trp. La secuencia que codifica el péptido guía es traducida inmediatamente después de su transcripción por un ribosoma que sigue de cerca a la ARN polimerasa a medida que avanza la transcripción.
|
Estructura secundaria de la región líder
|
|
Esquema de lo que sucede con niveles altos (b) y bajos (c) de triptófano |
Cuando las concentraciones de triptófano son altas, las concentraciones de ARNt cargados con triptófano también son altas. Esto permite que la traducción proceda rápidamente, pasando por los dos codones de Trp de las secuencia 1 hasta la secuencia 2 antes de que la secuencia 3 sea sintetizada por la ARN polimerasa. Ello provoca que la secuencia 2 quede cubierta por el ribosoma, lo que la inhabilita para aparearse con la secuencia 3 cuando esta es sintetizada; se forma la estructura atenuadora (secuencias 3 y 4) y la transcripción se interrumpe. En cambio, cuando las concentraciones de triptófano son bajas, el ribosoma se atasca en los dos codones de trp de la secuencia 1 porque hay menos ARNt cargado. La secuencia 2 por tanto, permanece libre mientras se sintetiza la 3, permitiendo que estas dos secuencias se apareen y que se produzca la transcripción. De esta manera, la proporción de transcritos que se atenúa desciende a medida que disminuye la concentración de triptófano.
|
Porción que se traduce de la región lider del operón triptófano
|
|
Porciónes que se traducen de las regiones líder de los operones de Fenilalanina (a) e Histidina (b) |
La atenuación hace bajar el nivel de triptófano 10 veces, pero si se combina con el mecanismo de represión puede llegar a producir un descenso de 600 veces el nivel.
El operón triptófano fue el primero en el que se descubrió el mecanismo de atenuación, pero no es el único en el que se da.
Otros ejemplos de represión por atenuación son:
Es obvio que en las células existen muchos genes que están sometidos a diferentes sistemas de regulación. Si nos fijamos en dos sistemas enzimáticos cualesquiera, puede darse los siguientes casos:
Esta situación puede explicarse suponiendo que los productos finales catalizados por los diferentes enzimas actúen como inductores o correpresores del otro sistema. Por ejemplo, el producto de un enzima induce la expresión de los genes de otro sistema enzimático, o por el contrario, el producto de otra enzima impide la expresión de los genes de otro sistema enzimático.
Por consiguiente, las interacciones entre operones son algo frecuente en los seres vivos y hacen más complejo el entendimiento de la regulación de la expresión génica.
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN FAGOS
Los bacteriófagos son virus que infectan a bacterias que han desarrollado a lo largo de la evolución mecanismos cada vez más eficientes para poner a su servicio la maquinaria de las células bacterianas a las que infectan, de forman que ponen a su servicio la síntesis de ADN y la síntesis de proteínas. Por tanto, el conocimiento profundo de los sucesos que tienen lugar durante la infección de una bacteria por un fago, nos indicará que genes del fago se están expresando para llevar a cabo su ataque y en qué momento lo hacen.
Los fagos han desarrollado diferentes sistemas para infectar a las bacterias. Los principales mecanismos utilizados por los fagos para dirigir la síntesis del ADN y de proteínas de la bacteria a la que infectan poniéndola a su servicio son los siguientes:
El fago ? posee ADN lineal doble hélice con extremos cohesivos. Cuando el fago ? infecta a E. coli pueden iniciarse dos respuestas diferentes:
Se pueden distinguir varias regiones en ADN lineal del fago ? en base al tipo o función de los genes que contiene cada una de ellas: regiones con funciones de regulación, funciones de integración (recombinación) del ADN del fago en el ADN bacteriano, regiones que contienen genes relacionados con la replicación del ADN, la lisis bacteriana, regiones con genes relacionados con la maduración del virus (regiones con genes para las proteínas de la cabeza y de la cola).
Mapa del Fago ? |
El mecanismo que utiliza el fago para expresar sus genes es la producción de proteínas activadoras o represoras de los operones del fago. Para explicar la regulación de este fago distinguiremos los siguientes aspectos:
Regulación durante el ciclo lítico
El ciclo lítico consta de tres fases sucesivas que se corresponden con tres rondas de transcripción:
En la transcripción temprano-inmediata la ARN polimerasa de E.coli se une al promotor de la hélice derecha (P R ) para transcribir hacia la derecha el gen cro y al promotor de la hélice izquierda (P L ) para transcribir la proteína N producto del gen N .
|
Transcripción temprano-inmediata de los genes N y cro |
El gen cro está relacionado con la decisión lisis versus lisogenia, por tanto, hablaremos más tarde de él.
La proteína N actúa como activador de la transcripción de los genes temprano-retrasados que son cII, O, P y Q en la hélice R que se transcribe hacia la derecha y del gen cIII en la hélice L que se transcribe hacia la izquierda. El producto N impide que termine la transcripción antes del gen Q interaccionando con la ARN polimerasa antes de que se inicie la transcripción cerca de los promotores P R y P L .
|
Transcripción temprano-retrasada |
Los genes cII y cIII están involucrados en la regulación de la lisogenia. Sin embargo, los genes O y P codifican para productos necesarios para la replicación del ADN. El producto del gen Q actúa como activador de la transcripción de los genes tardíos de manera semejante a como en el producto del gen N. La transcripción de los genes tardíos incluye los genes S y R que intervienen en la lisis bacteriana, los genes A, W, B, C, nu3, D, E FI y FII involucrados en la síntesis y el ensamblaje de las proteínas de la cabeza del fago, y los genes Z, U, V, G, T, H, M, L, H, I y J necesarios para la síntesis y ensamblaje de las proteínas de la cola del virus. De esta manera se completa el ciclo lítico del virus con la liberación de nuevas partículas virales, ya que se ha llevado a cabo la replicación del ADN, síntesis y ensamblaje de todas las proteínas necesarias (cabeza y cola), la lisis y maduración.
El producto N es muy inestable de manera que su concentración intracelular disminuye rápidamente, de forma que para que se establezca el ciclo lítico se necesita la expresión continua del gen N .
Regulación durante el ciclo lisogénico
Como ya hemos visto en el apartado anterior, una vez que el fago ha inyectado su ADN en el interior de la bacteria se produce la transcripción temprano- inmediata de los genes N y cro . Posteriormente el producto N estimula la transcripción de los genes cII en la hélice derecha (R) y del gen cIII en la hélice izquierda (L). Los productos cII y cIII estimulan la transcripción del gen cI y del gen rex a partir del promotor P RE (promotor para el establecimiento del represor). El gen cI codifica el represor I o represor del fago ? . El producto cIII protege a la proteína cII de la proteólisis. La proteína represora I bloquea las regiones operadoras O R y O L e impide la transcripción de los genes temprano-inmediatos N y cro , y por tanto, tampoco se expresan los genes O, P y Q (temprano-retrasados) ni los genes tardíos necesarios para el ciclo lítico. De esta forma se establece la lisogenia. Por tanto, el represor I impide el ciclo lítico.
|
Transcripción de los genes cI y rex a partir del promotor P RE |
Ahora bien, el establecimiento de la lisogenia necesita además que el ADN del fago se integre en el ADN bacteriano. Se ha visto que los productos cII y cIII estimulan la expresión de los genes necesarios para la integración a partir del promotor P INT situado en la región att-exo.
. |
Competencia entre lisis y lisogenia
La producción de la proteína represora I se puede llevar a cabo a partir de dos promotores diferentes. Como hemos visto la transcripción del gen cI se estimula por los productos cII y cIII a partir del promotor para el establecimiento del represor, P RE . El gen cI también se transcribir estimulado por el propio represor I a partir del promotor para el mantenimiento del represor, P RM . En este último caso, se trata de un proceso de autorregulación o regulación autógena , en el que una propia proteína ( represor I ) regula su propia expresión.
|
La integración y la escisión del ADN del fago son también procesos clave de la regulación del ciclo del fago ? . La integración del ADN circular del fago se produce por recombinación en la región att del ADN del virus y conduce a la aparición del profago (ADN integrado del virus). La situación opuesta consiste en la liberación o escisión del ADN del fago que vuelve a ser ADN doble hélice circular libre.
Se ha propuesto que la integración y la escisión se llevarían a cabo a través de una región de 15 pares de bases (región central O) común a las sedes att del virus y de la bacteria. La secuencia de estos quince pares de bases de la región O y los puntos de corte es la siguiente: GCTTT N TTTATA CTAA.
La integración requiere solamente la actuación de la proteína int , mientras que la escisión necesita de la actuación de las proteínas int y xis . Las proteínas cII y cIII son necesarias para producir altos niveles de la proteína int y para realizar una eficiente integración.
Se ha propuesto que la proteína int podría actuar como una endonucleasa de restricción que reconocería a la región O y produciría extremos cohesivos en el ADN el fago y de la bacteria.
Regulación simultánea de genes situados a distancia. Respuesta SOS.
La respuesta SOS se pone en marcha cuando hay grandes daños en el ADN.
Las proteínas que participan en la respuesta SOS se encuentran distribuidas en el genoma y su expresión se encuentra reprimida como consecuencia de la acción de un represor llamado lex A. La proteína lex A que está unida a regiones operadoras y bloquea la transcripción. Para que se induzca la expresión de estos genes es necesario que lex A se libere de las regiones operadoras, lo que ocurre cuando la proteína lex A se rompe de forma autoproteolítica, es decir, la propia lex A escinde un enlace peptídico entre Alanina y Glicina. Para que se estimule esta actividad autoproteolítica es necesario que lex A interaccione con otra proteína llamada RecA, que se une a ADN monohebra y en esas condiciones se une a lex A de modo que lex A se rompe. Como consecuencia de la ruptura se separa de las regiones operadoras y se permite la expresión de los genes que intervienen en la respuesta SOS y que están reprimidos.
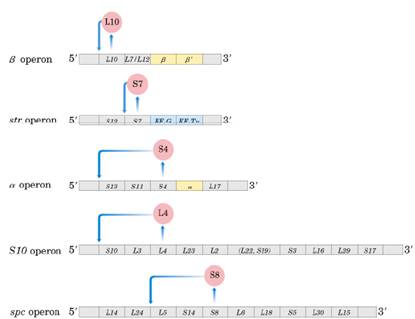
Síntesis coordinada de proteínas ribosómicas y ARNr:
Los ribosomas están constituidos por proteínas y ARNr. Estos dos componentes tendrán que expresarse de forma coordinada.
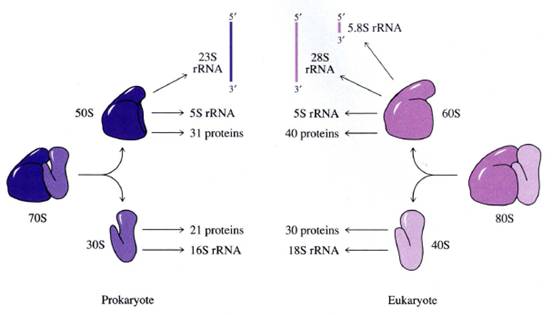
Regulación de la expresión de las proteínas (S o L, dependiendo de si son de la subunidad menor o mayor). Los genes que codifican para las proteínas ribosómicas se organizan en 20 operones. Su expresión se regula por un mecanismo llamado represión traduccional : lo que ocurre es que una de las proteínas que forma parte , es decir, que está codificada por el operón, cuando se expresa y está en exceso actúa como un represor de la traducción. El mecanismo por el que actúa es el siguiente: se une cerca del codón de inicio del ARNm que se ha transcrito a partir del operón. En este sitio el ARN es policistrónico. Se une a uno de los codones de inicio que suele estar al principio de este ARNm y bloquea la traducción.
Cada secuencia de una proteína tiene su codón de inicio. En principio solamente se debe bloquear la traducción de la región del codón en que se une (secuencia que codifica la proteína), pero se reprime todo porque el ARNm se encuentra formando una estructura en bucles. Para que se dé la traducción se tiene que romper esa estructura. La proteína lo que hace es estabilizar/favorecer esa estructura en bucles, de manera que bloquea la traducción de todo el ARNm porque hace que éste no pueda desplazarse y entrar en el ribosoma.
Proteína r represora
La regulación de la síntesis del ARNr se da mediante un mecanismo de adaptación que responde fundamentalmente a la presencia o ausencia de aminoácidos en el medio, y que se llama respuesta severa o estricta . Ésta se basa en que cuando hay pocos aminoácidos en el medio, el sitio A del ribosoma va a ser ocupado por un ARN de transferencia que estará descargado de aminoácidos. Como consecuencia se une una proteína al ribosoma que se llama factor de respuesta severa. Esta proteína cataliza la síntesis de guanosina tetrafosfato mediante la siguiente reacción:
La guanosina tetrafosfato se une a la ARN polimerasa y disminuye su afinidad por los promotores de los 7 operones en los que están codificados los ARNr. Este proceso es reversible, la guanosina tetrafosfato puede revertir hasta GTP, proceso llevado a cabo por el producto del gen spoT. Si no hay aminoácidos en el medio es inútil malgastar energía en sintetizar más ribosomas.
CREADO POR:
Marta Romero Pineda
Maribel Cano Linares